第 1 章 Base_R基础
1.1 基础拾遗
窗口可以通过拖动分割柱移动,也可以使用快捷键控制。比如:Ctrl+shift+1(2\3\4)
创建一个R脚本 ——Ctrl+shift+N
快速注释——Ctrl+shift+C
善于利用小标题实现功能分区(ctrl+shift+r)
ctrl+alt+r ——快速插入代码框
注释(Ctrl+shift+c); # ;
赋值(Alt+-): =, <- ;
函数的标志: ();
变量索引: $;
管道操作符: %>%, |>:读作 “然后” ;
比较或判断: >, < , >=, <=, ==, != ;
逻辑运算: &(与), |(或), !(非) ;
算数运算: +, -, *, /, ^, %%(取余), %/%(整除)。
1.2 数据基本操作
1.2.1 读取数据
mydata <- read_csv("data/data.csv") # 读取数据
# knitr::kable(mydata, align = "c")
colnames(mydata) # 查看表头## [1] "measure" "location" "sex" "age"
## [5] "cause" "metric" "year" "val"
## [9] "upper" "lower"
str(mydata) # 查看数据结构## spc_tbl_ [900 × 10] (S3: spec_tbl_df/tbl_df/tbl/data.frame)
## $ measure : chr [1:900] "Prevalence" "Prevalence" "Prevalence" "Prevalence" ...
## $ location: chr [1:900] "Global" "Global" "Global" "Global" ...
## $ sex : chr [1:900] "Male" "Female" "Both" "Male" ...
## $ age : chr [1:900] "All ages" "All ages" "All ages" "All ages" ...
## $ cause : chr [1:900] "HIV/AIDS" "HIV/AIDS" "HIV/AIDS" "HIV/AIDS" ...
## $ metric : chr [1:900] "Number" "Number" "Number" "Percent" ...
## $ year : num [1:900] 1990 1990 1990 1990 1990 1990 1990 1990 1990 1990 ...
## $ val : num [1:900] 3.87e+06 3.96e+06 7.83e+06 1.50e-03 1.53e-03 ...
## $ upper : num [1:900] 4.40e+06 4.55e+06 8.85e+06 1.71e-03 1.76e-03 ...
## $ lower : num [1:900] 3.35e+06 3.40e+06 6.88e+06 1.30e-03 1.32e-03 ...
## - attr(*, "spec")=
## .. cols(
## .. measure = col_character(),
## .. location = col_character(),
## .. sex = col_character(),
## .. age = col_character(),
## .. cause = col_character(),
## .. metric = col_character(),
## .. year = col_double(),
## .. val = col_double(),
## .. upper = col_double(),
## .. lower = col_double()
## .. )
## - attr(*, "problems")=<externalptr>
unique(mydata$cause) # 查看分类变量的取值(唯一值)## [1] "HIV/AIDS"
## [2] "Sexually transmitted infections excluding HIV"
# 其他查看数据的函数
# summary(mydata)
# head(mydata)
# tail(mydata)1.2.2 选择数据
## # A tibble: 15 × 10
## measure location sex age cause metric year
## <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 Prevalence Global Male All ag… HIV/… Number 2019
## 2 Prevalence Global Female All ag… HIV/… Number 2019
## 3 Prevalence Global Both All ag… HIV/… Number 2019
## 4 Prevalence Global Male All ag… HIV/… Perce… 2019
## 5 Prevalence Global Female All ag… HIV/… Perce… 2019
## 6 Prevalence Global Both All ag… HIV/… Perce… 2019
## # … with 9 more rows, and 3 more variables: val <dbl>,
## # upper <dbl>, lower <dbl>这里面出现了管道操作符号” |> “, 这个符号读作:然后。所以这个语句的意思是:首先,选中 mydata,然后,选择 cause 为”HIV/AIDS” 的行,然后,选择年份为 2019 年的行,最后,将选择好的数据赋值给 mydata2。
特别注意: = 是赋值符, == 为判断符。
1.2.3 保存数据
write.csv(mydata2, # 将 mydata2 这个数据
"HIV.csv") # 保存为 HIV.csv 文件1.3 数据类型与数据结构
1.3.1 常见的数据类型
R 语言常见数据类型有:数值类型(numeric),字符串(character),逻辑(logical),其他: POSIXct,POSIXt 等。
- 数值类型: 1, 2, 3, 4.5,3.8;
class(1)## [1] "numeric"
typeof(4.5)## [1] "double"- 字符串类型:“字符”, “123”, “ASIR”, “HIV”
class("123")## [1] "character"
typeof("ASIR")## [1] "character"- 逻辑: TRUE, FALSE, T, F;
class(T)## [1] "logical"
typeof(FALSE)## [1] "logical"1.3.2 特殊数据类型: NaN,Inf, NA, NULL
NaN:Not a number, 计算出错时候出现,比如 0/0,Inf/Inf
Inf : 无穷
NA:not availabel,缺失值。 NA 值具有传染性,任何数值与之发生关系均会变为 NA
NULL:空值。空值与 NA 的区别:比如一个教室稀稀拉拉坐了十几个学生,那么没有学生的位置可以视为缺失,而空值表示连座位也没有。
1.3.3 数据类型的转换
- numeric 与 logical 可以转化为 character,而 character 转化为 numeric 或者 logical 有可能出错,产生 NA 值。
a <- as.character(1990)
class(a)## [1] "character"
b <- as.numeric("1990")
class(b)## [1] "numeric"
c <- as.numeric("ABC")
## Warning: 强制改变过程中产生了NA
c## [1] NA- logical 可以转化为数值 0/1, 而在进行逻辑判断的时候, 0/1 也会被认为是 F/T
as.numeric(TRUE)## [1] 1
as.logical(1)## [1] TRUE1.3.4 数据结构
- 标量
单个元素组成的数据结构,比如”A”,123,TRUE 等。
### 向量
多个标量组成的一维的数据结构,比如 Vector。
# 生成向量
a <- c(1990:2019)
a## [1] 1990 1991 1992 1993 1994 1995 1996 1997 1998 1999
## [11] 2000 2001 2002 2003 2004 2005 2006 2007 2008 2009
## [21] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019
b <- c(1,2,3,4,5,6)
b## [1] 1 2 3 4 5 6
c <- rep("A",3) #repeat 前三个字母
c## [1] "A" "A" "A"
d <- seq(from=1,to=10,by=2) #sequence 前三个字母
d## [1] 1 3 5 7 9矩阵 (matrix),数据框 (data.frame), tipple
列表 (list) 与数组 (array)
1.4 作业
- 查看 data.csv 数据, 说出这个数据的性别有多少种?
data <- read_csv("data/data.csv")
unique(data$sex)## [1] "Male" "Female" "Both"- 从 data.csv 文档选择年龄标准化 患病率数据。
## # A tibble: 180 × 10
## measure locat…¹ sex age cause metric year val
## <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
## 1 Preval… Global Male Age-… HIV/… Rate 1990 148.
## 2 Preval… Global Fema… Age-… HIV/… Rate 1990 147.
## 3 Preval… Global Both Age-… HIV/… Rate 1990 147.
## 4 Preval… Global Male Age-… Sexu… Rate 1990 11293.
## 5 Preval… Global Fema… Age-… Sexu… Rate 1990 19439.
## 6 Preval… Global Both Age-… Sexu… Rate 1990 15387.
## # … with 174 more rows, 2 more variables: upper <dbl>,
## # lower <dbl>, and abbreviated variable name
## # ¹location- 从 data.csv 文档选择患病人数数据。
data |>
filter(metric == "Number")## # A tibble: 180 × 10
## measure locat…¹ sex age cause metric year val
## <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
## 1 Preval… Global Male All … HIV/… Number 1990 3.87e6
## 2 Preval… Global Fema… All … HIV/… Number 1990 3.96e6
## 3 Preval… Global Both All … HIV/… Number 1990 7.83e6
## 4 Preval… Global Male All … Sexu… Number 1990 2.71e8
## 5 Preval… Global Fema… All … Sexu… Number 1990 4.74e8
## 6 Preval… Global Both All … Sexu… Number 1990 7.46e8
## # … with 174 more rows, 2 more variables: upper <dbl>,
## # lower <dbl>, and abbreviated variable name
## # ¹location- 从 data.csv 文档中选择 1990 到 2010 年艾滋病的女性患病率数据,并写出为 HIV_female.csv 文档。
data |>
filter(year %in% c(1990:2010)) |>
filter(sex == "Female") |>
filter(cause == "HIV/AIDS") |>
filter(metric == "Rate") |>
write_csv("data/HIV_female.csv")- 从 data.csv 文档中选择 1990,1995,2000,2005,2010,2015年男性、女性其他传染性疾病患病率数据,并写出为 nonHIV.csv 文档
1.5 函数
1.5.1 函数的结构
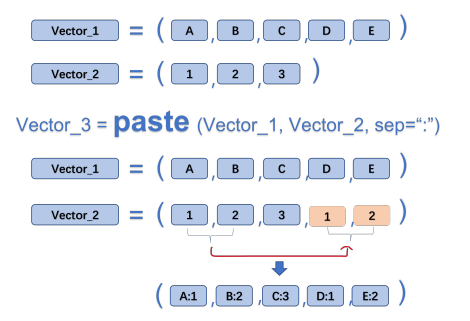
R 语言函数一般由三个部分构成:函数体(body),参数(formals) , 环境(environment)。可以通过相应的函数查看函数的相应部分。以常见的 “粘贴” 函数 paste() 为例:
# paste() 函数可以将两个向量对应的元素粘贴在一起。
a <- c("A","B","C")
b <- c(1:3)
c <- paste(a,b,sep = ":",collapse = NULL)
c## [1] "A:1" "B:2" "C:3"我们分别使用 body, formals, environment 函数查看 paste() 函数的相应部分。
# 查看函数结构
body(paste)## .Internal(paste(list(...), sep, collapse, recycle0))
# 查看函数参数
formals(paste)## $...
##
##
## $sep
## [1] " "
##
## $collapse
## NULL
##
## $recycle0
## [1] FALSE
# 查看函数来自哪个包
environment(paste)## <environment: namespace:base>1.5.2 函数的功能
函数就像一个加工厂,可以将 “原料” 通过一系列转变,然后输出相应的 “产品”
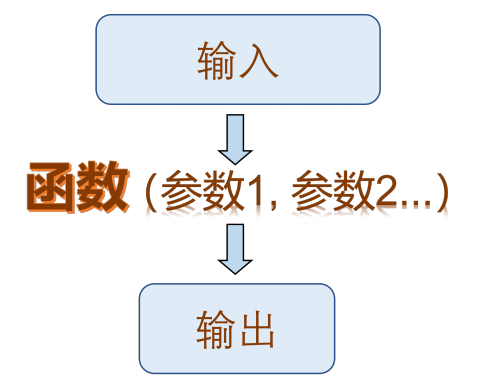
函数展示
这里的输入,可以是某个值、向量、 data.frame,或者是其他类型的数据;
输出,可以是数据,文档,图片等等。
1.5.3 函数的分类
R 语言的函数包括内置函数,外来函数(R 包),自编函数三大类。
- 实用的内置函数
# 查看R语言自带数据集
data()
# 描述性统计类
sum()
cumsum()
mean()
median()
sd()
quantile()
# 生成随机数
runif(n = , min = , max = ) # uniform,生成 n 个服从均匀分布的小数
round(runif(n=,min=,max=),# round() 函数空值小数点的位数
digits = 0)# 生成 n 个服从均匀分布的整数- 实用的外来函数(tidyverse)
# 根据变量取值进行筛选
filter()
# 选择变量
select()
# 生成变量
mutate()
# 排序
arrange()
# 分组统计
group_by() |>
summarize()- 演示(使用R自带数据)
## [1] "len" "supp" "dose"
# 查看数据结构
str(ToothGrowth)## 'data.frame': 60 obs. of 3 variables:
## $ len : num 4.2 11.5 7.3 5.8 6.4 10 11.2 11.2 5.2 7 ...
## $ supp: Factor w/ 2 levels "OJ","VC": 2 2 2 2 2 2 2 2 2 2 ...
## $ dose: num 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 ...
# 查看分组变量的取值
unique(ToothGrowth$supp)## [1] VC OJ
## Levels: OJ VC
# 生成变量(生成两个标签)
ToothGrowth <- ToothGrowth |>
mutate(剂量=ifelse(dose==0.5,"0.5mg",
ifelse(dose==1.0,"1.0mg","2.0mg"))) |>
mutate(补充喂养=ifelse(supp=="VC"," 维 C"," 橙汁"))
# 分组统计
summary_data <- ToothGrowth |>
group_by(补充喂养, 剂量) |>
summarize(
n=n(),
mean=mean(len),
sd=sd(len))- 自编函数
1.6 向量及其操作
向量为一系列标量的集合
1.6.2 向量的类型
- 字符串,数值, logical 类型
- 因子类型
R 语言有一类非常重要的变量类型,名为因子(factor)。因子可以视为分类变量的特殊类型,它既有值,又对值进行了排序(levels)。
# 如何生成因子
name1 <- c(0,1,2)
name2 <- factor(name1,
levels = c(0,1,2), #levels 必须与原始数据取值相同
labels = c("Male","Female","Both")) #labels 是为了
class(name1)## [1] "numeric"
class(name2)## [1] "factor"1.7 数据框及其操作
数据框是 R 语言最常用的二维表。
1.7.1 生成数据框
# 通过 read.csv 函数等读取
df <- read_csv()
# 通过向量组合生成
age <- c(20,30,18,26)
name <- c(" 赵"," 钱"," 孙"," 李")
score <- c(99,65,77,88)
df <- data.frame(age=age,
name=name,
score=score)1.7.2 数据框的下标索引
# 数据框下标索引中间有逗号,逗号前表示行,逗号后表示列 [row,col]
# 选择 df 前两列
df[,2] # 空着表示全选## [1] " 赵" " 钱" " 孙" " 李"
df[2,] # 选择 df 前两行## age name score
## 2 30 钱 65
df[2,2] # 选择 df 第二行第二列## [1] " 钱"
df[-3,] # 不选择第三行## age name score
## 1 20 赵 99
## 2 30 钱 65
## 4 26 李 88
df[c(1,3),2] # 选择 1, 3 行; 2 列## [1] " 赵" " 孙"
df[df$name==" 李",] # 选择姓李的数据## age name score
## 4 26 李 881.7.3 数据框如何生成新的列
# 比如生成身高数据
height <- c(178,180,169,175)
df$height <- height
df## age name score height
## 1 20 赵 99 178
## 2 30 钱 65 180
## 3 18 孙 77 169
## 4 26 李 88 1751.8 作业
- 查看函数帮助,说出 paste() 与 paste0() 的差别, sep 和 collapse 参数的作用。
?paste
?paste0
# paste 比 paste0() 多了一个参数: sepsep 和 collapse 参数的作用:
# 函数形式 paste (..., sep = " ", collapse =NULL)
# 示例
a <- c(1:4)
b <- c("A","B","C","D")
# sep 参数指定某个连接符,比如这里制定了下划线
paste(a,b,sep = "_",collapse = NULL)## [1] "1_A" "2_B" "3_C" "4_D"
# collapse 参数默认为 NULL,
# 如果是其他值,向量就会坍缩成一个字符串标量
paste(a,b,sep = "_",collapse = ":")## [1] "1_A:2_B:3_C:4_D"- 加载 R 内置数据 ToothGrowth, 按照喂养方式与剂量分组统计不同组牙齿生长长度的均值,标准差,中位数,四分位间距,最小值,最大值,以及老鼠的个数。
## 'data.frame': 60 obs. of 3 variables:
## $ len : num 4.2 11.5 7.3 5.8 6.4 10 11.2 11.2 5.2 7 ...
## $ supp: Factor w/ 2 levels "OJ","VC": 2 2 2 2 2 2 2 2 2 2 ...
## $ dose: num 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 ...
library(tidyverse)
# 分组统计
temp <- ToothGrowth |>
group_by(supp, dose) |>
summarise(len_mean = mean(len),
len_sd = round(sd(len), 1),
len_median = median(len),
len_IQR_lower = quantile(len, 0.25),
len_IQR_upper = quantile(len, 0.75),
len_min = min(len),
len_max = max(len),
mice_number = n())
knitr::kable(temp) # 展示数据| supp | dose | len_mean | len_sd | len_median | len_IQR_lower | len_IQR_upper | len_min | len_max | mice_number |
|---|---|---|---|---|---|---|---|---|---|
| OJ | 0.5 | 13.23 | 4.5 | 12.25 | 9.70 | 16.18 | 8.2 | 21.5 | 10 |
| OJ | 1.0 | 22.70 | 3.9 | 23.45 | 20.30 | 25.65 | 14.5 | 27.3 | 10 |
| OJ | 2.0 | 26.06 | 2.7 | 25.95 | 24.57 | 27.07 | 22.4 | 30.9 | 10 |
| VC | 0.5 | 7.98 | 2.7 | 7.15 | 5.95 | 10.90 | 4.2 | 11.5 | 10 |
| VC | 1.0 | 16.77 | 2.5 | 16.50 | 15.28 | 17.30 | 13.6 | 22.5 | 10 |
| VC | 2.0 | 26.14 | 4.8 | 25.95 | 23.38 | 28.80 | 18.5 | 33.9 | 10 |
- 生成 1 到 100 范围差值为 5 的等差序列 a,生成 1 到一百万倍数为 10 的等比序列 b。
# 等差序列
a <- seq(from = 1, to = 100, by = 5)
a## [1] 1 6 11 16 21 26 31 36 41 46 51 56 61 66 71 76 81
## [18] 86 91 96
# 等比序列
b <- 10^seq(from = 0, to = 6)
b## [1] 1e+00 1e+01 1e+02 1e+03 1e+04 1e+05 1e+06- 已知向量 a=c(1:18),b=c(5:36), 用两种方法找到只存在 a,b 某一个向量中的所有元素。
## 方法一
# duplicated() 函数判断一个向量每个元素是否重复,并生成一个逻辑向量
a <- c(1:18)
b <- c(5:36)
c <- c(a,b)#5:18 存在重复的元素
c <- c[!duplicated(c)]
c## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17
## [18] 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34
## [35] 35 36
## 方法二:使用 unique() 函数
# unique() 函数可以直接去重
a=c(1:18)
b=c(5:36)
c <- c(a,b)#5:18 存在重复的元素
c <- unique(c)
c## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17
## [18] 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34
## [35] 35 36
## 方法三:使用 setdiff() 函数:
# setdiff() 函数可以找到 a 或 b 互为补充的部分
a=c(1:18)
b=c(5:36)
# 1.
c <- setdiff(a,b)
c <- c(c,b)
c## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17
## [18] 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34
## [35] 35 36## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17
## [18] 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34
## [35] 35 36- 将 data.csv 的年龄,年份都设置为因子。
temp <- read_csv("data/data.csv")
# R 默认按照数值或字母先后顺序排序,因此这里不需要设定 levels
# 但是其他离散的分类变量,一定要检查顺序对不对,设定好 levels
temp$year <- factor(temp$year)
temp$age <- factor(temp$age)- 在 data.csv 中选择年龄标准化患病率数据,然后生成一列变量 se(标准误)。提示:根据正态性假设,标准误与区间存在以下关系: se=(区间上限-区间下限) /(2*1.96)。
## # A tibble: 360 × 11
## measure location sex age cause metric year
## <chr> <chr> <chr> <fct> <chr> <chr> <fct>
## 1 Prevalence Global Male Age-st… HIV/… Perce… 1990
## 2 Prevalence Global Female Age-st… HIV/… Perce… 1990
## 3 Prevalence Global Both Age-st… HIV/… Perce… 1990
## 4 Prevalence Global Male Age-st… HIV/… Rate 1990
## 5 Prevalence Global Female Age-st… HIV/… Rate 1990
## 6 Prevalence Global Both Age-st… HIV/… Rate 1990
## # … with 354 more rows, and 4 more variables:
## # val <dbl>, upper <dbl>, lower <dbl>, se <dbl>- 在 data.csv 中生成年龄组变量(5 年间隔),如果年份在 1990 年到 1994年之间,生成 year2 为”year:1990-94”;如果年份在 1995 年到 1999 年之间,生成”year:1995-99”, 以此类推。提示:使用 ifelse(test,yes,no) 函数。
## 方法一,用 ifelse:
temp |>
mutate(year2 = ifelse(year %in% c(1990:1994),"year:1990_94",
ifelse(year%in%c(1995:1999),"year:1995_99",
ifelse(year%in%c(2000:2004),"year:2000_04",
ifelse(year%in%c(2005:2009),"year:2005_09",
ifelse(year%in%c(2010:2014),"year:2010_14",
"year:2015_19"))))))## # A tibble: 900 × 11
## measure location sex age cause metric year
## <chr> <chr> <chr> <fct> <chr> <chr> <fct>
## 1 Prevalence Global Male All ag… HIV/… Number 1990
## 2 Prevalence Global Female All ag… HIV/… Number 1990
## 3 Prevalence Global Both All ag… HIV/… Number 1990
## 4 Prevalence Global Male All ag… HIV/… Perce… 1990
## 5 Prevalence Global Female All ag… HIV/… Perce… 1990
## 6 Prevalence Global Both All ag… HIV/… Perce… 1990
## # … with 894 more rows, and 4 more variables:
## # val <dbl>, upper <dbl>, lower <dbl>, year2 <chr>
## 方法二,用 case_when:
temp <- temp |>
mutate(year2=case_when(year%in%c(1990:1994)~"year:1990_94",
year%in%c(1995:1999)~"year:1995_99",
year%in%c(2000:2004)~"year:2000_04",
year%in%c(2005:2009)~"year:2005_09",
year%in%c(2010:2014)~"year:2010_14",
year%in%c(2015:2019)~"year:2015_19"))
####### 正常计算OR #############
# 患病 不患病
#暴露 80 30
#非暴露 20 70
# 患病 不患病
#暴露 0.8 0.3
#非暴露 0.2 0.7
# 患病暴露比(odds) 0.8/0.2
# 不患病暴露比(odds) 0.3/0.7
# 计算OR值 (0.8/0.2)/(0.3/0.7)=9.33
#### 转换成logisttic计算 #######
# y患病=1,不患病=0
# x暴露=1,不暴露=0
## 由于是二分类变量 1-p(y=1)=p(y=0)
# 患病 不患病
#暴露 p(y=1) p(y=0)
#非暴露 p'(y=1) p'(y=0)
## (1)OR = [p(y=1)/p'(y=1)]/[(p(y=0)/p'(y=0))]=[p(y=1)/p(y=0)]/[p'(y=1)/p'(y=0)]
## 构建logistic方程 ln [P(y=1)/(1-P(y=1))] = bx+d
## 推出P(y=1)/(1-P(y=1))=P(y=1)/P(y=0)=exp(b*x+d)
## (2)暴露组 p(y=1)/p(y=0) = exp(b*1+d)
## (3)非暴露组 p'(y=1)/p'(y=0) = exp(b*0+d)=exp(d)
## 根据(1)推导代入(2)(3)得到
## OR=exp(b*1+d)/exp(d)=exp(b)
########### R语言模拟程序 ###########
x <- c(rep(1,80),rep(0,20),rep(1,30),rep(0,70))
y <- rep(c(1,0),c(100,100))
df <- data.frame(x,y)
df$y <- factor(df$y,c(0,1))
df$x <- factor(df$x,c(0,1))
df2 <- df
names(df2) <- c("暴露","患病")
table(df2)## 患病
## 暴露 0 1
## 0 70 20
## 1 30 80
(0.8/0.2)/(0.3/0.7)## [1] 9.333
table(df$x,df$y)##
## 0 1
## 0 70 20
## 1 30 80##
## Call: glm(formula = y ~ x, family = binomial(), data = df)
##
## Coefficients:
## (Intercept) x1
## -1.25 2.23
##
## Degrees of Freedom: 199 Total (i.e. Null); 198 Residual
## Null Deviance: 277
## Residual Deviance: 224 AIC: 228
log(9.333)## [1] 2.234